宏基因组数据分析第一步:质量控制
厨师拿到原材料之后要进行处理,我们拿到测序公司给的下机测序数据也不能直接就拿来分析。
质量控制(Quality Control)的目的是从原始数据中过滤出我们想要分析的数据。
哪些序列需要被过滤呢?
要去除的序列
不合格的reads不是好reads
单个碱基的质量
常用的二代测序核心原理是“边合成边测序”,测序需要DNA聚合酶的催化。随着测序过程进行,DNA聚合酶的活性会下降,错误匹配的碱基会被添加到待测的模板链上。二代测序的另一个特点是大规模的平行测序,说人话就是一条模板链会被测非常多次。假如一条序列被测了10000次,对于这条链上第N个位置上的碱基A,有9900次测得是A,其余100次的结果是G/T/C。那么这个位置的碱基正确率就是:
$$正确率=\frac{正确测序次数}{总测序次数}=\frac{9900}{10000}=0.99$$
错误率表示为:
$$错误率=\frac{错误测序次数}{总测序次数}=\frac{100}{10000}=0.01$$
我们从测序公司手上得到的一般是压缩后的fastq文件,常见文件后缀为fastq.gz,fq.gz。
我们可以用这条命令查看压缩包内文件的格式,而不用解压数据(解压后的fastq文件非常大!)
zcat SRR17228757_1.fastq.gz | head -n 4 #表示查看SRR17228757_1.fastq.gz前4行的内容,也就是第一条序列。
@SRR17228757.1 1 length=301
AGTTCCACTCCTACGGGAGGCAGCAGTGGGGAATATTGGACAATGGGCGAAAGCCTGATCCAGCAATGCCGCGTGAGTGATGAAGGCCTTAGGGTTGTAAAGCTCTTTTACCCGGGATGATAATGACAGTACCGGGAGAATAAGCTCCGGCTAACTCCGTGCCAGCAGCCGCGGTAATACGGAGGGAGCTAGCGTTTTTCGGAAATACTGGGCGTAAAGCGCACGTAGGCGGCGCAGCAAGTCAGGGGTGAAATCCCGGGGCTCAACCCCGGAACTGCCTCTGAAAATGCCGTGCTCTTCG
+SRR17228757.1 1 length=301
GGGGGGGGGGGGGGGGGGGGGGGGGGGGGGCEGGGGGGF9CECFCFFGEGGGDGGGF,CFGGGGGGGDFECEGCGGGGGGGD99<,4EEFGGGGGGDFE9,C<EGGFGEFFAFFCGGGGGGGGGDAFG?GGGGGGG7BEFGGDGGGGGDFFECCFGFGGGGGGGGFG7FFEGGG@:>C9DCC<@7<=<:CFG:*=F*<CCEGGGGGCFG9:*2::EGG7+*:?EC8*;EDEGGGCC>ECFDGF?9*875:;CEG7C+8C*?5*2<C6*9CED*:C<F9C98+<EFG+<<++2*/11#####
根据 illumina公司官网对FASTQ格式介绍 ,每一条序列有4行数据,依次代表序列的标识符、序列内容、分隔符和碱基质量评分。我们作为用户要关心的主要是第二行和第四行的内容。
@SRR17228757.1 1 length=301 #序列标识符
AGTTCCACTCCTAC***CGTGCTCTTCG #序列内容,太长用*省略
+SRR17228757.1 1 length=301 #分隔符
GGGGGGGGGGGGGG***+2*/11##### #碱基质量评分,太长用*省略
你肯定会好奇为什么要用这些英文字母和符号来表示每个碱基的质量分数,为什么不能直接用数字来表示呢?因为碱基质量分数Q的定义是这样的:
$$\textit{Q}=-10\log_{10}{e} $$
其中e是某个位置碱基的错误率。碱基错误率越低,Q的值越高。假如某个碱基的错误率e值为0.0001,那么对应的Q值即为40。如果碱基错误率e值为1,对应的Q值为0。FASTQ文件本质上是一个文本文件,我们当然可以用两个数字来存储每个碱基的质量评分,就像这样
@test_sequence 1
AGTTCCACTCCTACGG
+test_sequence 1
23453234313243241233575430572345 #碱基得分乱写的,仅作演示
虽然跟上面真实的FASTQ文件看起来很怪,但还是表达了我们需要的信息。计算机也能够读懂。但当我们得到的序列数量达到百万、千万甚至上亿条时,FASTQ文件的大小会很恐怖。为了节约大家电脑/服务器的储存空间,科学家们决定设计一套规则来转换Q值。
Phred编码
计算机上的字母、符号、数字都会消耗计算机的存储空间。最早出现的编码规则,
ASCii编码规则
中,每个英文字母(不分大小写)、数字还有一些其他符号都占据一个字节。所以如果用两个数字来表示Q评分就需要2个字节,而如果把Q评分和ASCii编码中的符号对应起来就能节省一半空间。
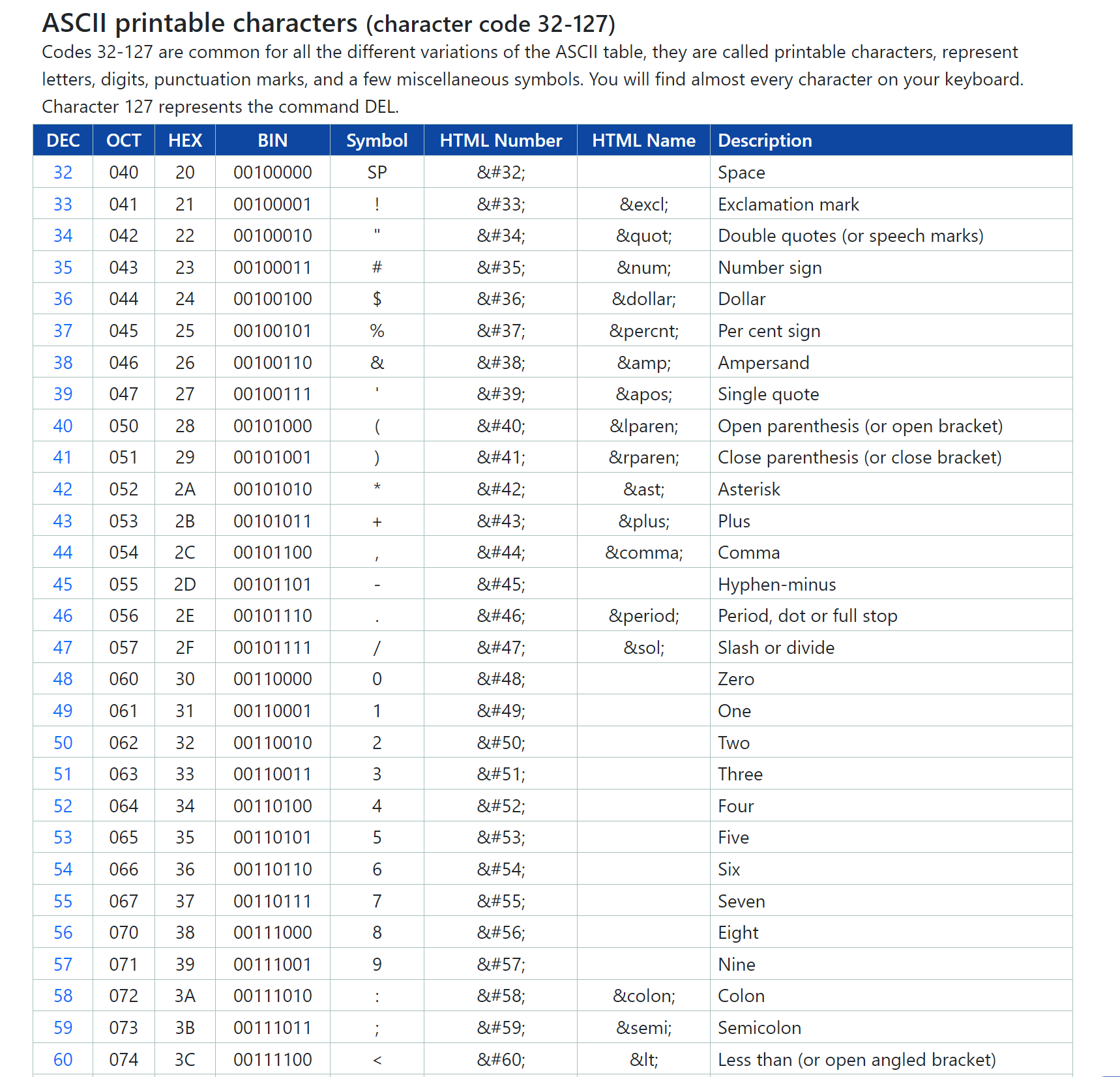
观察发现,能让人看见的第一个ASCii编码的字符是感叹号“!”,对应的十进制编码是33。如果我们用“!”来对应Q0,那么Q20对应5(DEC 53),Q30对应?(DEC 63),Q40对应I(DEC 73)。最后一个人可见的字符是~(DEC 126)。因此ASCII编码中第33-126的所有字符都可以用来表示碱基质量得分,这也就是最初的Sanger Phred+33编码。使用Phred+33编码时,碱基质量得分最小为0(DEC 33),最大为93(DEC 126)。
提示
如果对FASTQ的编码感兴趣,可以阅读这篇文章和维基百科对FASTQ格式的介绍
Cock P J A, Fields C J, Goto N, et al. The Sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants[J]. Nucleic acids research, 2010, 38(6): 1767-1771.
我想告诉你的是,这些测序机构搞出了很多编码格式,如Phred+33, Phred+64, Solexa+64。所以我们首先要搞明白我们得到的FASTQ文件是什么编码。我知道网上有很多脚本可以帮我们做到,但是我们想找到一个更简单的方法,也就是用其他人写好的、特别是发表到期刊上的软件来告诉我们。
使用vsearch的–fastq_chars参数来得到碱基质量分数编码
VSEARCH是一款开源免费的宏基因组分析工具。该工具是另一常用软件USEARCH的开源替代,因为USEARCH的源代码不公开,且64位版本的软件要收费。64位的USEARCH价格是885美元😨。我们作为穷鬼一定要大力支持USEARCH😄。
vsearch的下载方式在其 Github项目界面 写得非常详细,我这里下载的是Debian 2.22.1-1版本
sudo apt install vsearch
vsearch --version #vsearch v2.22.1_linux_x86_64, 7.7GB RAM, 16 cores
使用man vsearch查看vsearch手册并用/--fastq_chars定位参数说明
--fastq_chars filenameSummarize the composition of sequence and quality strings contained in the input FASTQ file. For each of the four DNA letters, –fastq_chars gives the number of occurrences of the letter, its relative frequency and the length of the longest run of that letter. For each character present in the quality strings, –fastq_chars gives the ASCII value of the character, its relative frequency, and the number of times a k-mer of that character appears at the end of quality strings. The length of the k-mer can be set using –fastq_tail (4 by default). The command –fastq_chars tries to automatically detect the quality encoding (Solexa, Illumina 1.3+, Illumina 1.5+ or Illumina 1.8+/Sanger) by analyzing the range of observed quality score values. In case of success, –fastq_chars suggests values for the –fastq_ascii (33 or 64), –fastq_qmin and –fastq_qmax options to be used with the other commands that require a FASTQ input file.
根据上述说明,vsearch可以告诉我们碱基质量编码到底是Solexa+64,Phred+64还是Phred+33。
让我们来实操一下,你也可以随便找一个fastq文件(vsearch不支持fastq.gz文件)。你可以用这个命令来提取你压缩文件中的一些序列来进行验证,从而避免解压它们:
zcat original_file.fastq.gz | head -n N > output_file.fastq
这条命令的意思是查看original_file.fastq.gz文件前N行,并把这前N行的内容重定向到output_file.fastq。如果我们想查看前M条序列,那么N=4*M。
coding@Debian:~$ zcat SRR17228757_1.fastq.gz | head -n 40000 > 10k.fastq
coding@Debian:~$ vsearch --fastq_chars 10k.fastq
vsearch v2.22.1_linux_x86_64, 7.7GB RAM, 16 cores
https://github.com/torognes/vsearch
Reading FASTQ file 100%
Read 10000 sequences.
Qmin 35, QMax 71, Range 37
Guess: -fastq_qmin 2 -fastq_qmax 38 -fastq_ascii 33
Guess: Original Sanger format (phred+33)
Letter N Freq MaxRun
------ ---------- ------ ------
A 711667 23.6% 6
C 724653 24.1% 8
G 997734 33.1% 11
T 575946 19.1% 6
Char ASCII Freq Tails
---- ----- ------ ----------
'#' 35 1.7% 10000
')' 41 0.1% 0
'*' 42 1.3% 0
'+' 43 0.7% 0
',' 44 0.2% 0
'-' 45 0.0% 0
'.' 46 0.0% 0
'/' 47 0.2% 0
'0' 48 0.2% 0
'1' 49 0.1% 0
'2' 50 0.3% 0
'3' 51 0.2% 0
'4' 52 0.1% 0
'5' 53 0.7% 0
'6' 54 0.3% 0
'7' 55 0.8% 0
'8' 56 1.0% 0
'9' 57 0.6% 0
':' 58 1.2% 0
';' 59 0.4% 0
'<' 60 1.1% 0
'=' 61 0.4% 0
'>' 62 0.6% 0
'?' 63 0.8% 0
'@' 64 0.8% 0
'A' 65 0.4% 0
'B' 66 0.3% 0
'C' 67 5.6% 0
'D' 68 2.0% 0
'E' 69 5.1% 0
'F' 70 8.9% 0
'G' 71 63.9% 0
vsearch检查了10000条序列,告诉我们这是Phred+33编码的FASTQ文件。
一条序列的质量
我们已经知道了单个碱基的质量分数的定义、储存方式。接下来我们要看如何表示一条序列的质量。这里我们需要用到专门的质量控制(Quality Control,QC)软件。目前最常用的软件是由Babraham Institute开发的 FastQC 。我还想给大家推荐一款中国人开发的软件 FASTP 。
FastQC
参照 官方文档 进行安装,这里我用的是Debian 12进行安装。
警告
请注意,最新版的fastqc版本号为0.12.1[2023-05-01发布]。如果你使用的是Debian12或者Ubuntu22.04,软件仓库中的fastqc版本为0.11.9[2020-01-08发布]。我们这里要下载0.12.1版本的fastqc。
# install default Java Runtime Environment (JRE) on Debian/Ubuntu
coding@Debian:~$ sudo apt install default-jre
...
done.
Setting up default-jre-headless (2:1.17-74) ...
Setting up openjdk-17-jre:amd64 (17.0.12+7-2~deb12u1) ...
Setting up default-jre (2:1.17-74) ...
Processing triggers for libgdk-pixbuf-2.0-0:amd64 (2.42.10+dfsg-1+deb12u1) ...
# check java version if jre is successfully installed.
coding@Debian:~$ java -version
openjdk version "17.0.12" 2024-07-16
OpenJDK Runtime Environment (build 17.0.12+7-Debian-2deb12u1)
OpenJDK 64-Bit Server VM (build 17.0.12+7-Debian-2deb12u1, mixed mode, sharing)
# search fastqc in the apt repository
coding@Debian:~$ sudo apt search fastqc
Sorting... Done
Full Text Search... Done
atropos/stable 1.1.31+dfsg-3+b3 amd64
NGS read trimming tool that is specific, sensitive, and speedy
fastqc/stable 0.11.9+dfsg-6 all
quality control for high throughput sequence data
# download fastqc v0.12.1 from official website
coding@Debian:~$ cd .softwares/
coding@Debian:~/.softwares$ URL=https://www.bioinformatics.babraham.ac.uk/projects/fastqc/fastqc_v0.12.1.zip
coding@Debian:~/.softwares$ wget -c $URL
coding@Debian:~/.softwares$ sudo apt install zip unzip
...
Unpacking zip (3.0-13) ...
Setting up unzip (6.0-28) ...
Setting up zip (3.0-13) ...
coding@Debian:~/.softwares$ unzip fastqc_v0.12.1.zip
coding@Debian:~/.softwares$ cd FastQC/ && ls
cisd-jhdf5.jar fastqc_icon.ico INSTALL.txt LICENSE_JHDF5.txt org RELEASE_NOTES.txt uk
Configuration Help jbzip2-0.9.jar LICENSE.txt README.md run_fastqc.bat
fastqc htsjdk.jar LICENSE net README.txt Templates
# add the path of fastqc to ~/.bashrc
# add this sentence to the file
coding@Debian:~/.softwares/FastQC$ sudo vim ~/.bashrc
export PATH="$PATH:$HOME/.softwares/FastQC"
coding@Debian:~/.softwares/FastQC$ source ~/.bashrc
# check the version of our installed fastqc
coding@Debian:~$ fastqc --version
FastQC v0.12.1
使用fastqc --help可以查看fastqc的详细用法,我们作为使用者需要设置的参数是-o --outdir (指定输出目录)和-t --threads (使用计算机的线程数)。
-o --outdir Create all output files in the specified output directory.
Please note that this directory must exist as the program
will not create it. If this option is not set then the
output file for each sequence file is created in the same
directory as the sequence file which was processed.
-t --threads Specifies the number of files which can be processed
simultaneously. Each thread will be allocated 250MB of
memory so you shouldn't run more threads than your
available memory will cope with, and not more than
6 threads on a 32 bit machine
提示
如何查看自己电脑上有多少个可用的线程?
方法一
coding@Debian:~$ cat /proc/cpuinfo| grep "processor"| wc -l
16 #我这里使用的是8核心,16线程的Intel i5-13400F
方法二
coding@Debian:~$ sudo apt install neofetch
...
After this operation, 137 MB of additional disk space will be used.
Do you want to continue? [Y/n] y
...
Processing triggers for libc-bin (2.36-9+deb12u4) ...
Processing triggers for fontconfig (2.14.1-4) ...
Processing triggers for hicolor-icon-theme (0.17-2) ...
我们使用FastQC来检查我们手上的序列。这里使用的序列来自一篇Nature Communications上的文章。
Wang, N., Wang, T., Chen, Y. et al. Microbiome convergence enables siderophore-secreting-rhizobacteria to improve iron nutrition and yield of peanut intercropped with maize. Nat Commun 15, 839 (2024). https://doi.org/10.1038/s41467-024-45207-0
16S 扩增子序列数据被作者上传到了NCBI SRA数据库(BioProject PRJNA788265)。 我这里使用sratoolkit下载了编号为SRR17228757的测序数据,并用fasterq-dump拆分成双端数据。
# create a folder for fastqc results
coding@Debian:~$ mkdir fastqc_results
coding@Debian:~$ fastqc SRR17228757_1.fastq.gz SRR17228757_2.fastq.gz --outdir ./fastqc_results --threads 16
Started analysis of SRR17228757_1.fastq.gz
...
Started analysis of SRR17228757_2.fastq.gz
...
Analysis complete for SRR17228757_1.fastq.gz
...
Analysis complete for SRR17228757_2.fastq.gz
coding@Debian:~$ cd fastqc_results/
coding@Debian:~/fastqc_results$ ls
SRR17228757_1_fastqc.html SRR17228757_1_fastqc.zip SRR17228757_2_fastqc.html SRR17228757_2_fastqc.zip
fastqc生成了两个html文件和两个zip压缩文件。我们直接用浏览器打开这两个html文件。
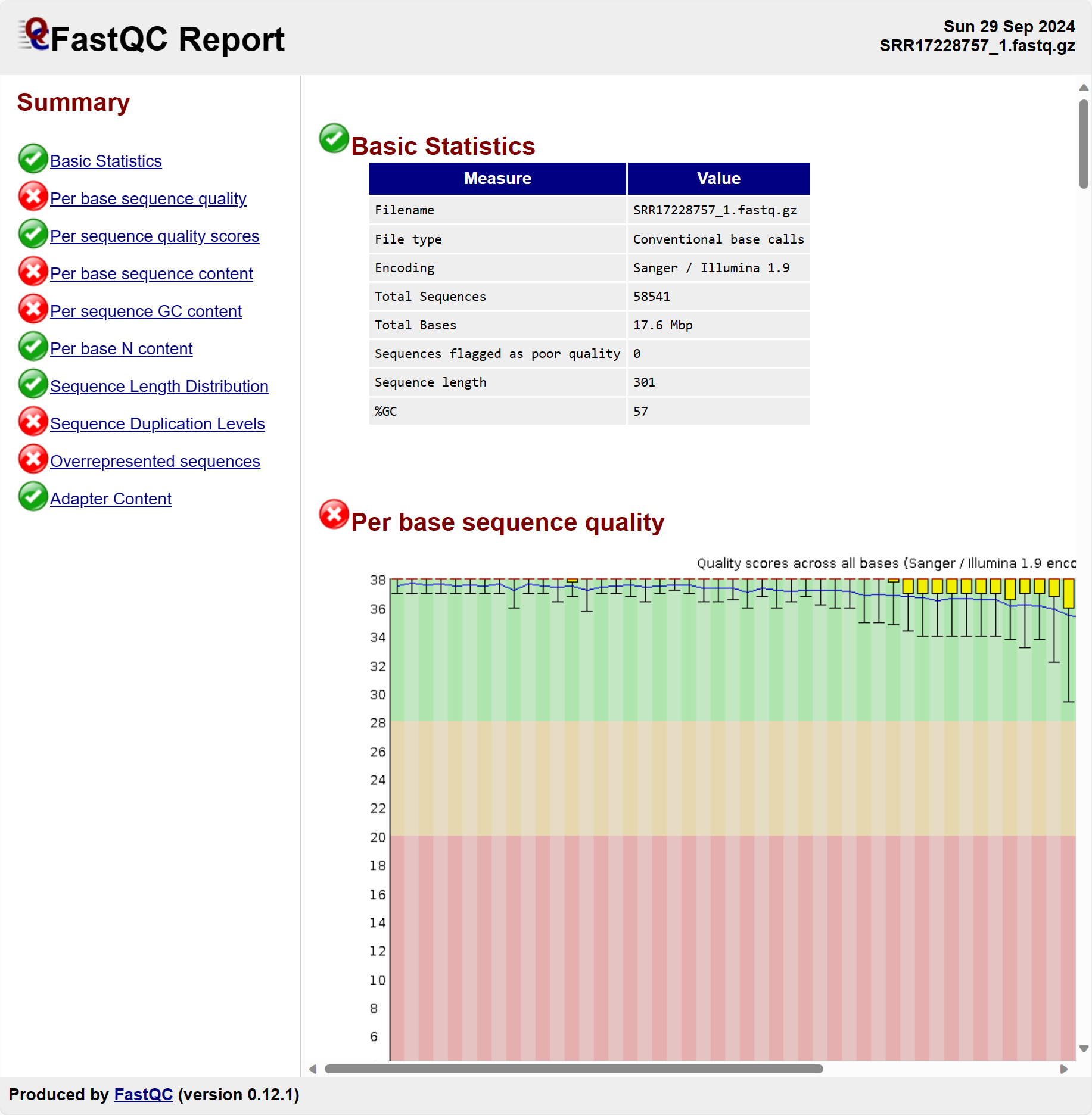
关于FastQC各个部分的解释,网上有很多中文教程介绍。对于研究生来讲,最好的学习方式是去看FastQC的 官方英文说明 。
Basic Statistics
| Measure | Value | 解释 |
|---|---|---|
| Filename | SRR17228757_1.fastq.gz | 被QC的文件名 |
| File type | Conventional base calls | 文件内容 |
| Encoding | Sanger / Illumina 1.9 | 碱基质量分数的编码格式 |
| Total Sequences | 58541 | 包含的序列条数 |
| Total Bases | 17.6 Mbp | 包含的碱基总数 |
| Sequence flagged as poor quality | 0 | 低质量序列 |
| Sequence length | 301 | 每条序列长度 |
| %GC | 57 | 所有序列的GC百分比 |
注释
这里有一些注意点
- 1. FastQC自动帮我们算出了碱基质量分数的编码格式,为Sanger / Illumina 1.9。Illumina 1.9是Illumina公司测序数据处理pipeline的版本号。
2. 碱基总数的单位是Mbp,意为Megabase pair, million base pair。
> 3. 测序公司说的xxGb实际上指的是他们能测得的碱基总数是多少个gigabase pair (多少billion base pairs)
Per Base sequence quality
Per Sequence quality scores
Per Base Sequence Content
Per Sequence GC Content
Per Base N Content
Sequence Length Distribution
Sequence Duplication Levels
Overrepresented Sequences
Adapter Content
FASTP
FASTP的 Github文档 写得很详细。读者可以选择用conda安装,我这里选择安装作者编译好的二进制文件。
# create a folder for our downloaded softwares
coding@Debian:~$ mkdir .softwares
# move into .softwares directory
coding@Debian:~$ cd .softwares/
# download the latest version
coding@Debian:~/.softwares$ wget -c http://opengene.org/fastp/fastp
# add executable permission to fastp
coding@Debian:~/.softwares$ chmod a+x ./fastp
在~/.bashrc文件末尾添加fastp文件的路径
coding@Debian:~$ sudo vim ~/.bashrc
# add this sentence to the last of the file
export PATH="$PATH:$HOME/.softwares"
# exit and refresh
收了adapter“50w”的reads
来自“境外势力”的reads
References
illumina: FASTQ files explained: https://knowledge.illumina.com/software/general/software-general-reference_material-list/000002211
illumina: Measuring sequencing accuracy https://sapac.illumina.com/science/technology/next-generation-sequencing/plan-experiments/quality-scores.html
ASCII Table https://www.ascii-code.com/
Cock P J A, Fields C J, Goto N, et al. The Sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants[J]. Nucleic acids research, 2010, 38(6): 1767-1771. https://doi.org/10.1093/nar/gkp1137
Wikipedia. FASTQ format https://en.wikipedia.org/wiki/FASTQ_format
Rognes T, Flouri T, Nichols B, et al. VSEARCH: a versatile open source tool for metagenomics[J]. PeerJ, 2016, 4: e2584.
Babraham Bioinformatics. FastQC. https://www.bioinformatics.babraham.ac.uk/projects/fastqc/
Shifu Chen. 2023. Ultrafast one-pass FASTQ data preprocessing, quality control, and deduplication using fastp. iMeta 2: e107. https://doi.org/10.1002/imt2.107